Bilaga 1 PM: Rekommendationer för återrapportering av konstitutionella varianter från cancergenetiska utredningar inom klinisk patologi
Detta PM är framtaget av den nationella arbetsgruppen för ärftlig cancer vid RCC i samverkan (hädanefter ”NAG”). NAG har uppmärksammat att ett ökande antal analyser utförda inom klinisk patologi på vuxna patienter kan ge information om ärftligt ökad risk för cancersjukdom, och att det efterfrågas stöd om i vilka situationer och för vilka genetiska varianter som återrapportering kan anses vara kliniskt relevant för onkogenetisk uppföljning.
NAG har särskilt granskat det nationella arbetet inom Genomic Medicine Sweden, där man inom arbetsgruppen för solida tumörer (GMS-ST) har tagit fram en genpanel benämnd GMS560 avsedd att användas som ett diagnostiskt verktyg vid ett flertal cancerdiagnoser. En mellan orterna varierande andel av analyserna kommer utföras som så kallade parade analyser, vilket innebär att tumör- och normalvävnad (typiskt DNA från blodprov) analyseras parallellt. Vid en sådan analys kan man omedelbart få kännedom om konstitutionella (medfödda, oftast ärftliga) genetiska varianter som kan ha betydelse för riskprediktion, uppföljning, riskreducerande åtgärder och testning av familjemedlemmar.
Detta PM är indelat i två delar, en bakgrund med allmänna överväganden inför återrapportering av konstitutionella varianter, följt av en konkret rekommendation avseende vilka gener som är aktuella att återrapportera vid olika vanligt förekommande tumörformer. Sist i PM:et finns en tabell med en översikt över NAG:s rekommendationer. Ett viktigt syfte med tabellen är avgränsning, dvs att medfödda varianter i gener som inte återfinns i tabellen inte ska återrapporteras rutinmässigt.
Kort sammanfattning av NAG:s rekommendationer
- Detta PM ska inte tolkas som att NAG rekommenderar någon specifik utredning, syftet är endast att beskriva principer för hur fynd som uppkommer vid utredning av vuxna patienter med cancersjukdom inom klinisk patologi bör hanteras ur ett ärftlighetsperspektiv.
- Ur ärftlighetsperspektiv rekommenderas parad analys.
- Om endast tumöranalys utförts så bör status som konstitutionell variant fastställas innan återrapportering som ett potentiellt relevant ärftligt fynd. NAG rekommenderar laboratorier att följa rekommendationer från ESMO:s arbetsgrupp avseende vilka somatiskt detekterade varianter som (i avsaknad av parad analys) bör leda till kompletterande konstitutionell utredning, med viss modifiering enligt detta PM.
- Endast sannolikt patogena (klass 4) och patogena (klass 5) varianter bör svaras ut. Varianter av oklar klinisk signifikans (VUS, klass 3) bör i typfallet registreras i laboratoriets databas men inte rapporteras kliniskt.
- Principer för genetisk vägledning inför bred diagnostisk testning behöver etableras på regional nivå. NAG har erfarenhet av liknande processer och deltar gärna i sådana diskussioner.
- Utöver formell klassificering av genetiska varianter så har NAG även övervägt den kliniska relevansen (”clinical actionability”) av fynd i olika gener och vid olika diagnoser. Utgående från detta rekommenderar NAG att för vuxna patienter med cancersjukdom så ”bör” eller ”kan” fastställda konstitutionella patogena eller sannolikt patogena varianter i definierade gener rapporteras ut antingen vid samtliga cancerutredningar, alternativt med specifikationer för olika tumörtyper. Dessa rekommendationer sammanfattas i en tabell i slutet av detta PM.
Bakgrund
Parad eller icke-parad analys
I det följande diskuteras endast fastställda konstitutionella varianter, något som kan erhållas vid parad genetisk analys. Även om NAG ur ett ärftlighetsperspektiv rekommenderar parad analys så förutses att diagnostiska analyser inom klinisk patologi initialt kommer att ske som icke-parad analys vid flera orter (d.v.s. endast tumören analyseras). Tumörfynd bör inte återrapporteras som ett potentiellt relevant ärftligt fynd för klinisk handläggning, utan i dessa fall bör status som konstitutionell variant först verifieras (diskutera gärna med kliniskt genetiskt laboratorium vid behov).
För att minimera merarbetet efter icke-parad analys rekommenderar NAG att laboratorier vid klinisk patologi övergripande förhåller sig till principer som publicerats av European Society of Medical Oncology (ESMO) Precision Medicine Working Group (PMWG) år 2023, baserat på data från Memorial Sloan Kettering [1]. NAG anser dock att en något mer konservativ hållning än ESMO PMWG är aktuell, och att därför endast varianter i gener som diskuteras senare i detta PM rutinmässigt bör återrapporteras. Specifikt menar NAG till skillnad från ESMO PMWG att det är prematurt att rutinmässigt eftersöka konstitutionella varianter i RET-genen. Dels är sådana fynd förhållandevis ovanliga (0,6 promille i aktuell publikation [1]), dels föreligger delvis osäkra siffror avseende cancerrisk (penetrans), se tex Loveday et al, 2018 [2].
Vidare bör nämnas att man vid icke-parad analys behöver ta hänsyn till det som ESMO PMWG benämner ”germline conversion rate” (GCR), vilket kan uttydas som sannolikheten att en tumörpåvisad variant visar sig vara av konstitutionellt ursprung. Vid mycket låg GCR är NAG enig med ESMO PMWG om att det inte är rimligt att gå vidare med kompletterande blodprov inom rutindiagnostik. Av gener som diskuteras i detta PM har följande en GCR <5% enligt ESMO:s publikation och bör därför inte leda till rutinmässig ärftlighetsutredning efter tumörfynd vid icke-parad analys: APC, CDH1, CDKN2A, PTEN, STK11 och TP53. ESMO PMWG anser dock att uppföljande ärftlighetsanalys bör ske efter tumörfynd av APC, PTEN och TP53 om patienten insjuknat före 30 års ålder.
Genetisk vägledning
En patient har rätt till information innan en genetisk utredning, rätt att få ställa frågor till vårdpersonal, och även rätt att avstå från en planerad utredning. Vid rutinutredning i samband med cancerdiagnos måste systemet för genetisk vägledning vara så enkelt som möjligt, men frågan får inte glömmas bort. NAG har varit delaktig i arbetet i några regioner där så kallade ”snabbspår” för utredning av ärftlighet för cancer hos vuxna har upprättats med stöd av respektive RCC, och kan rekommendera att en liknande struktur införs för den breda cancerdiagnostiska genetiska utredningen.
En erfarenhet från snabbspåren är att det är viktigt med förankring och utbildningsinsatser i de lokala teamen som praktiskt handlägger patienterna, där samtliga i teamet bör involveras i processen och skriftliga riktlinjer för hur handläggningen skall ske utformas. Erfarenheten är också att det räcker med kortare muntlig information kompletterad med skriftlig information till patienten där patienten hänvisas till kontaktsjuksköterska vid frågor, samt att det finns genetisk expertis tillgänglig i de fåtal fall där ytterligare diskussion önskas innan analysen utförs. Efter analys hanteras normalsvar av behandlande läkare, och patienter med påvisad konstitutionell patogen variant i relevant gen remitteras till onkogenetisk mottagning för fortsatt omhändertagande.
Fynd vid pediatriska cancerformer (barn) kräver särskild omsorg och behandlas inte i detta PM.
”Clinical actionability”
I arbetet kring att definiera vilka konstitutionella genetiska varianter som är relevanta att återrapportera i olika kontext så har det svåröversatta begreppet ”clinical actionability” lanserats, främst inom ClinGen-nätverket [11, 12]. Begreppet innefattar en flerdimensionell matris som leder till en samlad bedömning av klinisk relevans, där faktorer som måste beaktas inkluderar hur allvarligt hälsotillstånd som är kopplat till det genetiska fyndet, hur hög sannolikheten är för sjukdom (penetrans, expressivitet), hur effektiva medicinska interventioner bedöms vara, vilka hälsorisker som är kopplade till aktuella interventioner, och avslutningsvis evidensgraden för de olika delbedömningarna. Sådana genomgångar utgör bland annat grunden för den lista av ”sekundära fynd” som ACMG har upprättat [13], ett antal gener som man inom amerikansk sjukvård anser bör svaras ut oavsett vilken klinisk indikation som har föranlett genetisk analys.
I europeiska sammanhang har man förhållit sig avvaktande till att rapportera sekundära fynd [14], och NAG avser inte att här ta ställning till denna fråga. Vi vill emellertid påstå att resonemanget kring sekundära fynd inte är helt relevant i kontexten av diagnostiska cancergenetiska analyser, då samtliga utvalda gener har koppling till den kliniska frågeställningen om cancersjukdom. Det generella resonemanget kring ”clinical actionability” är dock applicerbart, då evidensen för klinisk nytta avgör hur ofta och i vilka specifika kliniska situationer som en variant bör rapporteras.
Utgående från dessa allmänna resonemang, samt från den erfarenhet som NAG har inhämtat genom att vara delaktig i utformningen av de olika nationella vårdprogrammen för cancersjukdom (särskilt kapitlen kring ärftlighet), vill NAG lämna följande konkreta rekommendationer avseende återrapportering av konstitutionella patogena varianter för onkogenetisk handläggning. Baserat på kunskap och erfarenhet inom NAG graderas rekommendationerna i två nivåer, varianter som bör utrapporteras och varianter som kan utrapporteras.
Rekommendationer
Observera att i det följande diskuteras endast handläggning av påvisade konstitutionella varianter. Vid icke-parad tumöranalys, se resonemang ovan avseende fynd i generna APC, CDH1, CDKN2A, PTEN, STK11 och TP53.
Genetiska fynd som bör svaras ut vid samtliga cancerutredningar
För ett fåtal välbeskrivna gener anser NAG att det finns så god evidens för uppenbar klinisk nytta mätt som reducerad mortalitet och morbiditet att fynd av konstitutionella patogena varianter i dessa gener bör utrapporteras till beslutsförmögna vuxna individer vid samtliga diagnostiska cancerutredningar.
Inom ClinGens ”clinical actionability”-arbete har följande cancerassocierade gener vid PM:ets färdigställande i september 2024 den högsta konsensusklassificeringen ”Definitive Actionability” [16]: APC, BRCA1, BRCA2, EPCAM, MLH1, MSH2 och MSH6. För EPCAM bör förtydligas att endast 3’ deletionen (som påverkar MSH2) är kliniskt relevant vid onkogenetisk utredning. Utöver dessa gener anser NAG att även fynd av två varianter i genen MUTYH (ClinGen ”Strong Actionability”) kan svaras ut vid alla cancerutredningar, då även MUTYH-relaterad autosomalt recessivt ärftlig polypos har god evidens för nyttan av förebyggande åtgärder. Detta utgör en konservativ lista, och samtliga åtta gener rekommenderas att svaras ut som sekundära fynd enligt ACMG [13], Genomics England [16, 17] och French Society of Predictive and Personalized Medicine (SFMPP) [18].
Som tillägg till ovanstående kliniska argument så visar även hälsoekonomiska modeller att germline-testning av bland annat generna BRCA1, BRCA2, MLH1 och MSH2 i en oselekterad (yngre) population kan vara mer kostnadseffektivt än den nuvarande kriteriebaserade testningen [19, 20]. Även om situationen med tumörtestning inte är direkt jämförbar med populationstestning så bedömer NAG att det sannolikt skulle vara hälsoekonomiskt försvarbart att rapportera ut fynd i dessa gener vid samtliga diagnostiska cancerutredningar.
Genetiska fynd vid bröstcancer
Vid bröstcancer bör konstitutionella patogena varianter i generna BRCA1, BRCA2 och PALB2 svaras ut (se även det nationella vårdprogrammet).
TP53 utgör ett specialfall. Genen är den mest frekvent somatiskt muterade genen i ett flertal cancerformer och konstitutionella patogena varianter är mycket ovanliga (se resonemang ovan om handläggning efter icke-parad analys och fynd i tumörvävnad). Dessutom är det välbeskrivet att fynd av TP53-varianter i blodprov kan utgöras av så kallade CHIP (Clonal Hematopoiesis of Indeterminate Potential), ofta subklonalt men CHIP har även påvisats som orsak i en förhållandevis hög andel vid en variantallelfrekvens som talar för heterozygot förekomst [21]. Slutsatsen av detta är att i avsaknad av påvisad segregation i en familj så bör fynd av en patogen TP53-variant i ett blodprov alltid bekräftas i en annan vävnad, t.ex. uppodlade fibroblaster, innan varianten hanteras kliniskt som ett konstitutionellt fynd [22]. På grund av dessa omständigheter rekommenderar också det europeiska referensnätverket GENTURIS att för kvinnor utan familjehistoria bör TP53-analys begränsas till bröstcancer vid 45 år eller yngre [23]. Sammantaget anser NAG därför vid parad analys att konstitutionella patogena varianter i TP53 bör eftersökas endast vid bröstcancer ≤ 45 år och endast rapporteras efter att fyndet bekräftats i annan vävnad än blodprov.
Vid bröstcancer kan även proteintrunkerande konstitutionella patogena varianter i generna ATM, BARD1, CHEK2, RAD51C och RAD51D svaras ut. Skälet till den svagare rekommendationen är att bröstcancerrisken vid patogena varianter i dessa gener är lägre (särskilt vid rutinanalys utan misstänkt ärftlighet) och att effekten av förebyggande åtgärder inte har lika hög evidensgrad som för de gener som har bör-rekommendationen. Den huvudsakliga patogena mekanismen för dessa gener är funktionsförlust (”loss of function”, LOF) och enligt nuvarande kunskapsläge är proteintrunkerande varianter som grupp associerade med en högre cancerrisk än missensevarianter [24]. Därför rekommenderas tills vidare att endast konstitutionella patogena trunkerande varianter (frameshift, nonsense, splicepåverkande varianter eller större strukturella varianter) rapporteras vidare.
Notera att RAD51C och RAD51D har den högre klassningen bör vid äggstockscancer nedan.
Genetiska fynd vid epitelial äggstockscancer
Vid epitelial äggstockscancer bör konstitutionella patogena varianter i generna BRCA1, BRCA2, BRIP1, EPCAM (endast 3’ deletionen), MLH1, MSH2, MSH6, PALB2, RAD51C och RAD51D svaras ut (se även det nationella vårdprogrammet). Avseende BRIP1, RAD51C och RAD51D så gäller detta endast för proteintrunkerande varianter (se resonemang under bröstcancer ovan).
Vid epitelial äggstockscancer kan även konstitutionella patogena varianter i genen PMS2 svaras ut. Skälet till den svagare rekommendationen för PMS2 är dels att cancerrisken är lägre än för andra Lynchs syndrom-associerade gener (särskilt vid rutinanalys utan misstänkt ärftlighet), dels att analys av PMS2 försvåras på grund av pseudogensproblematik, vilket kräver särskild kompetens av utförande laboratorium.
Genetiska fynd vid prostatacancer
Vid prostatacancer bör konstitutionella patogena varianter i genen BRCA2 svaras ut (se även det nationella vårdprogrammet).
NAG menar att det inte finns tillräcklig evidens för att rutinmässigt svara ut konstitutionella patogena varianter i andra gener än BRCA2 av ärftlighetsskäl vid prostatacancerutredning. I vårdprogrammet rekommenderas också testning av BRCA1 av behandlingsprediktiva skäl. NAG tar inte i detta PM upp behandlingsprediktiva aspekter, och prostatacancerrisken vid BRCA1 bedöms för låg för att motivera onkogenetisk rekommendation specifikt vid denna diagnos. Notera dock att BRCA1 ingår i den grupp av gener som NAG anser bör svaras ut vid samtliga cancerdiagnoser.
Genetiska fynd vid tjock- och ändtarmscancer
Vid kolorektalcancer bör konstitutionella patogena varianter i generna APC, BMPR1A, EPCAM (endast 3’ deletionen), MLH1, MSH2, MSH6, POLD1, POLE, PTEN, SMAD4 och STK11 svaras ut (se även det nationella vårdprogrammet).
Fynd av två konstitutionella patogena varianter (homozygota eller sammansatt heterozygota) i genen MUTYH bör också svaras ut, då MUTYH associeras med ett autosomalt recessivt ärftligt polypostillstånd. Man bör fastställa att varianterna är belägna på varsin kromosom (i trans), men det kan inte anses vara ett krav inom rutinpatologin utan kan utföras via onkogenetisk mottagning. Observera dock att heterozygot bärarskap (fynd av endast en patogen variant) inte bör rapporteras ut rutinmässigt.
Vid kolorektalcancer kan även konstitutionella patogena varianter i genen PMS2 svaras ut. Skälet till den svagare rekommendationen för PMS2 är dels att cancerrisken är lägre än för andra Lynchs syndrom-associerade gener (särskilt vid rutinanalys utan misstänkt ärftlighet), dels att analys av PMS2 försvåras på grund av pseudogensproblematik, vilket kräver särskild kompetens av utförande laboratorium.
Genetiska fynd vid livmoderkroppscancer
Vid livmoderkroppscancer bör konstitutionella patogena varianter i generna EPCAM (endast 3’ deletionen), MLH1, MSH2, MSH6, och PTEN svaras ut (se även det nationella vårdprogrammet).
Vid livmoderkroppscancer kan även konstitutionella patogena varianter i genen PMS2 svaras ut. Skälet till den svagare rekommendationen för PMS2 är dels att cancerrisken är lägre än för andra Lynchs syndrom-associerade gener (särskilt vid rutinanalys utan misstänkt ärftlighet), dels att analys av PMS2 försvåras på grund av pseudogensproblematik, vilket kräver särskild kompetens av utförande laboratorium.
Genetiska fynd vid malignt melanom
Vid melanom bör konstitutionella patogena varianter i genen CDKN2A svaras ut (se även det nationella vårdprogrammet).
Vid melanom kan även konstitutionella patogena varianter i generna BAP1 och CDK4 svaras ut (se fördjupat resonemang i bilaga 1 till det nationella vårdprogrammet).
Genetiska fynd vid magsäckscancer
Vid diffus ventrikelcancer bör konstitutionella patogena varianter i generna CDH1 och CTNNA1 svaras ut (se även det nationella vårdprogrammet samt PM på sfmg.se). NAG har noterat att CTNNA1 inte är med på nuvarande design för genpanelen GMS560.
Genetiska fynd vid bukspottkörtelcancer
Vid pankreascancer kan konstitutionella patogena varianter i generna CDKN2A, PRSS1, och STK11 svaras ut (se även det nationella vårdprogrammet). Patogena varianter i dessa tre gener är förknippade med en tydligt förhöjd risk för bukspottkörtelcancer, men anledningen till att NAG anger den svagare rekommendationen kan är att evidensen för tydlig nytta av kontrollprogram och profylaktisk kirurgi fortfarande är förhållandevis svaga. Observera också att analys av PRSS1 försvåras på grund av pseudogensproblematik, vilket kräver särskild kompetens av utförande laboratorium.
Övriga gener och diagnoser
NAG har inga rekommendationer om rutinmässig rapportering av konstitutionella fynd utöver för de gener och diagnoser som beskrivs ovan. I individuella fall där det bedöms relevant att rapportera andra resultat bör fynd diskuteras med klinisk genetik eller annan onkogenetisk expertis innan konstitutionella fynd svaras ut.
Dokumentversion och uppdateringar
Detta PM utgör version 2.0, daterat 2024-09-10. NAG planerar att revidera dokumentet återkommande vid behov, och kolleger inom svensk sjukvård är välkomna att kontakta NAG om man önskar att särskilda diagnoser och/eller gener belyses ytterligare.
Innehåll i tabellen
Sist i detta PM finns en tabell som sammanfattar de rekommendationer som ges i PM:et. Diagnosspecifika fynd av patogen variant i gener som bör rapporteras ut är markerade med grön färg, diagnosspecifika fynd som kan rapporteras ut är markerade med grå färg, och fynd i gen som bör rapporteras ut vid samtliga cancerformer hos vuxen individ är markerade med blå färg.
Medlemmar i arbetsgruppen
För aktuella medlemmar i NAG ärftlig cancer, se sidan Cancergenetik på sfmg.se.
Vid publiceringen av detta PM utgjordes NAG ärftlig cancer av följande personer:
Umeå: Anna Rosén, Christina Edwinsdotter Ardnor
Uppsala: Ylva Paulsson-Karlsson, Anna Poluha, Stefanos Tsiaprazis
Örebro: Aniruddh Kashyap
Stockholm: Emma Tham, Svetlana Lagercrantz
Linköping: Anna-Lotta Hallbeck, Ekaterina Kuchinskaya
Göteborg: Fredrik Persson, Sara Orrsjö, Theofanis Zagoras
Lund: Marie Stenmark-Askmalm, Hans Ehrencrona
Referenser
- Kuzbari Z, et al. Germline-focused analysis of tumour-detected variants in 49,264 cancer patients: ESMO Precision Medicine Working Group recommendations. Ann Oncol. 2023 Mar;34(3):215-227. doi: 10.1016/j.annonc.2022.12.003.
- Loveday C, et al. p.Val804Met, the Most Frequent Pathogenic Mutation in RET, Confers a Very Low Lifetime Risk of Medullary Thyroid Cancer. J Clin Endocrinol Metab. 2018 Nov 1;103(11):4275-4282. doi: 10.1210/jc.2017-02529.
- Richards S, et al. Standards and guidelines for the interpretation of sequence variants: a joint consensus recommendation of the American College of Medical Genetics and Genomics and the Association for Molecular Pathology. Genet Med. 2015 May;17(5):405-24. doi: 10.1038/gim.2015.30.
- https://cspec.genome.network/cspec/ui/svi/
- Zion TN, et al. Clinical validity assessment of genes for inclusion in multi-gene panel testing: A systematic approach. Mol Genet Genomic Med. 2019 May;7(5):e630. doi: 10.1002/mgg3.630.
- Eccles DM, et al. BRCA1 and BRCA2 genetic testing-pitfalls and recommendations for managing variants of uncertain clinical significance. Ann Oncol. 2015 Oct;26(10):2057-65. doi: 10.1093/annonc/mdv278.
- Macklin SK, et al. Physician interpretation of variants of uncertain significance. Fam Cancer.2019 Jan;18(1):121-126. doi: 10.1007/s10689-018-0086-2.
- Welsh JL, et al. Clinical Decision-Making in Patients with Variant of Uncertain Significance in BRCA1 or BRCA2 Genes. Ann Surg Oncol. 2017 Oct;24(10):3067-3072. doi: 10.1245/s10434-017-5959-3.
- Mersch J, et al. Prevalence of Variant Reclassification Following Hereditary Cancer Genetic Testing. JAMA. 2018 Sep 25;320(12):1266-1274. doi: 10.1001/jama.2018.13152.
- Esterling L, et al. Impact of a Cancer Gene Variant Reclassification Program Over a 20-Year Period. JCO Precis Oncol. 2020 Aug 27;4:PO.20.00020. doi: 10.1200/PO.20.00020.
- Hunter JE, et al. A standardized, evidence-based protocol to assess clinical actionability of genetic disorders associated with genomic variation. Genet Med. 2016 Dec;18(12):1258-1268. doi: 10.1038/gim.2016.40.
- Berg JS, et al. A semiquantitative metric for evaluating clinical actionability of incidental or secondary findings from genome-scale sequencing. Genet Med. 2016 May;18(5):467-75. doi: 10.1038/gim.2015.104.
- Miller DT, et al. ACMG SF v3.1 list for reporting of secondary findings in clinical exome and genome sequencing: A policy statement of the American College of Medical Genetics and Genomics (ACMG). Genet Med. 2022 Jul;24(7):1407-1414. doi: 10.1016/j.gim.2022.04.006.
- de Wert G, et al. Opportunistic genomic screening. Recommendations of the European Society of Human Genetics. Eur J Hum Genet. 2021 Mar;29(3):365-377. doi: 10.1038/s41431-020-00758-w.
- https://search.clinicalgenome.org/kb/downloads#section_actionability
- Turnbull C, et al. Cancer genetics, precision prevention and a call to action. Nat Genet. 2018 Sep;50(9):1212-1218. doi: 10.1038/s41588-018-0202-0. Epub 2018 Aug 29. Erratum in: Nat Genet. 2019 Jan;51(1):196.
- https://www.genomicsengland.co.uk/initiatives/100000-genomes-project/additional-findings
- Pujol P, et al. Guidelines for reporting secondary findings of genome sequencing in cancer genes: the SFMPP recommendations. Eur J Hum Genet. 2018 Dec;26(12):1732-1742. doi: 10.1038/s41431-018-0224-1.
- Manchanda R, et al. Cost-effectiveness of Population-Based BRCA1, BRCA2, RAD51C, RAD51D, BRIP1, PALB2 Mutation Testing in Unselected General Population Women. J Natl Cancer Inst. 2018 Jul 1;110(7):714-725. doi: 10.1093/jnci/djx265.
- Zhang L, et al. Population genomic screening of all young adults in a health-care system: a cost-effectiveness analysis. Genet Med. 2019 Sep;21(9):1958-1968. doi: 10.1038/s41436-019-0457-6.
- Coffee B, et al. A substantial proportion of apparently heterozygous TP53 pathogenic variants detected with a next-generation sequencing hereditary pan-cancer panel are acquired somatically. Hum Mutat. 2020 Jan;41(1):203-211. doi: 10.1002/humu.23910.
- Daly MB, et al. NCCN Clinical Practice Guidelines in Oncology: Genetic/Familial High-Risk Assessment: Breast, Ovarian, and Pancreatic, version 1.2023. Available for download from NCCN.org.
- Frebourg T, et al; European Reference Network GENTURIS. Guidelines for the Li-Fraumeni and heritable TP53-related cancer syndromes. Eur J Hum Genet. 2020 Oct;28(10):1379-1386. doi: 10.1038/s41431-020-0638-4.
- Breast Cancer Association Consortium, et al. Breast Cancer Risk Genes - Association Analysis in More than 113,000 Women. N Engl J Med. 2021 Feb 4;384(5):428-439. doi: 10.1056/NEJMoa1913
Bilaga 1. Tabell med sammanfattning av rekommendationer i PM
Diagnosspecifika fynd av patogen variant i gener som bör rapporteras ut är markerade med grön färg, diagnosspecifika fynd som kan rapporteras är markerade med grå färg, och fynd i gen som bör rapporteras ut vid samtliga cancerformer hos vuxen individ är markerade med blå färg. Se PM för detaljer.
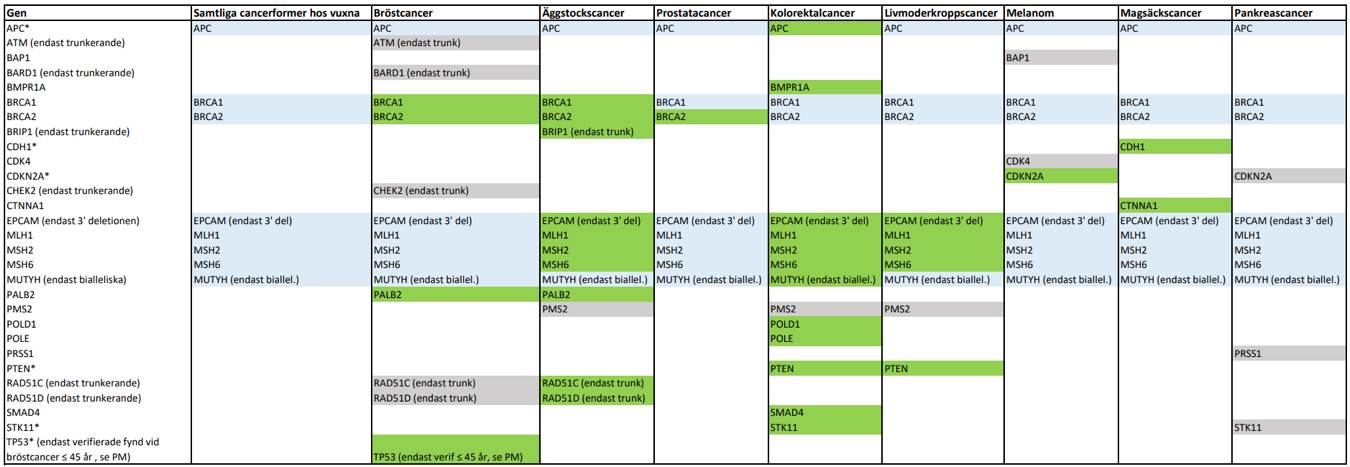
* Vid icke-parad tumöranalys bör noteras att APC, CDH1, CDKN2A, PTEN, STK11 och TP53 har en mycket låg "germline conversion rate", se fördjupad diskussion i PM.
